原始教程链接:https://github.com/iMetaScience/iMetaPlot/tree/main/NMDS
写在前面
非度量多维尺度分析(Non-metric multidimensional scaling, NMDS),是基于相异矩阵或距离矩阵进行排序分析的间接梯度分析方法,在微生物组研究中可以用来展示群落beta多样性。本期我们挑选2022年2月24日刊登在iMeta上的Linking soil fungi to bacterial community assembly in arid ecosystems - iMeta:西农韦革宏团队焦硕等-土壤真菌驱动细菌群落的构建,选择文章的Figure 6C进行复现,基于vegan包,讲解和探讨和NMDS分析和可视化的方法,先上原图:
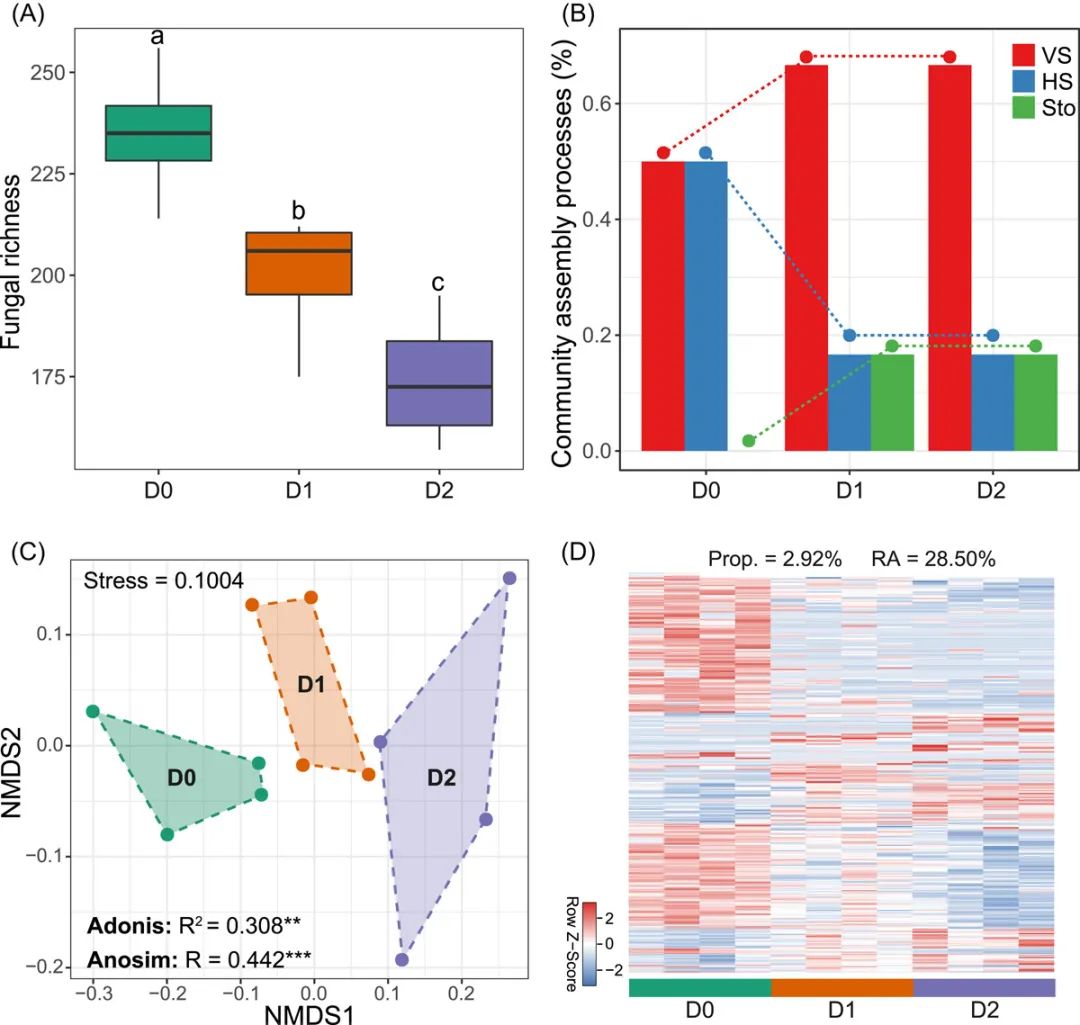
接下来,我们将通过详尽的代码逐步拆解原图,最终实现对原图的复现。代码编写及注释:农心生信工作室。
R包检测和安装
01
安装核心R包vegan以及ggplot2,并载入所有R包。
if (!require("vegan")) install.packages('vegan') if (!require("ggplot2")) install.packages('ggplot2') # 加载包 library(vegan) library(ggplot2)讯享网
生成测试数据
02
由于缺少原始数据,因此本例使用vegan包自带的dune数据集进行测试。dune包含了20个样品,每个样品有30个物种丰度,每一行是一个样品,每一列是一个物种。
讯享网# 载入dune数据集 data(dune) #载入dune包含分组信息等的元数据(即metadata),分组信息为Management列 data(dune.env)
NMDS分析
03
获取数据后,即可利用vegan包进行NMDS分析。
#计算bray_curtis距离 distance <- vegdist(dune, method = 'bray') #NMDS排序分析,k = 2预设两个排序轴 nmds <- metaMDS(distance, k = 2) #> Run 0 stress 0. #> Run 1 stress 0. #> ... Procrustes: rmse 4.e-05 max resid 0.000 #> ... Similar to previous best #> Run 2 stress 0. #> ... New best solution #> ... Procrustes: rmse 0.0 max resid 0.0 #> Run 3 stress 0. #> ... New best solution #> ... Procrustes: rmse 1.e-05 max resid 5.57604e-05 #> ... Similar to previous best #> Run 4 stress 0. #> Run 5 stress 0. #> Run 6 stress 0. #> ... New best solution #> ... Procrustes: rmse 5.e-06 max resid 1.e-05 #> ... Similar to previous best #> Run 7 stress 0. #> Run 8 stress 0. #> Run 9 stress 0. #> Run 10 stress 0. #> Run 11 stress 0. #> Run 12 stress 0. #> Run 13 stress 0. #> Run 14 stress 0. #> ... Procrustes: rmse 5.e-06 max resid 1.e-05 #> ... Similar to previous best #> Run 15 stress 0. #> Run 16 stress 0. #> Run 17 stress 0. #> ... Procrustes: rmse 3.001088e-06 max resid 9.e-06 #> ... Similar to previous best #> Run 18 stress 0. #> Run 19 stress 0. #> Run 20 stress 0. #> ... Procrustes: rmse 2.027412e-05 max resid 6.e-05 #> ... Similar to previous best #> * Best solution repeated 4 times #查看结果 #summary(nmds)04
获取可视化所需数据。
讯享网#获得应力值(stress) stress <- nmds$stress #将绘图数据转化为数据框 df <- as.data.frame(nmds$points) #与分组数据合并 df <- cbind(df, dune.env)
NMDS可视化
05
根据分组绘制一个最基础的散点图。
p <- ggplot(df, aes(MDS1, MDS2))+ geom_point(aes(color = Management), size = 5)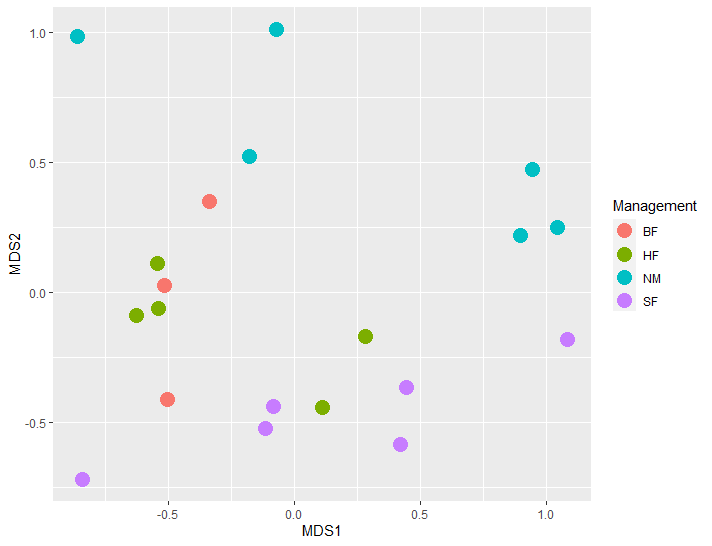
06

我们注意到,原图中,每个分组被连接成不规则的多边形并用不同颜色表示,我们可以通过ggplot2中geom_polygon()来绘制。geom_polygon()会按照数据中出现的顺序连接观测值,内部可填充颜色。
讯享网p <- ggplot(df, aes(MDS1, MDS2))+ geom_point(aes(color = Management), size = 5)+ geom_polygon(aes(x = MDS1, y = MDS2, fill = Management, group = Management, color = Management), alpha = 0.3, linetype = "longdash", linewidth = 1.5) #通过按顺序连接观测值绘制多边形
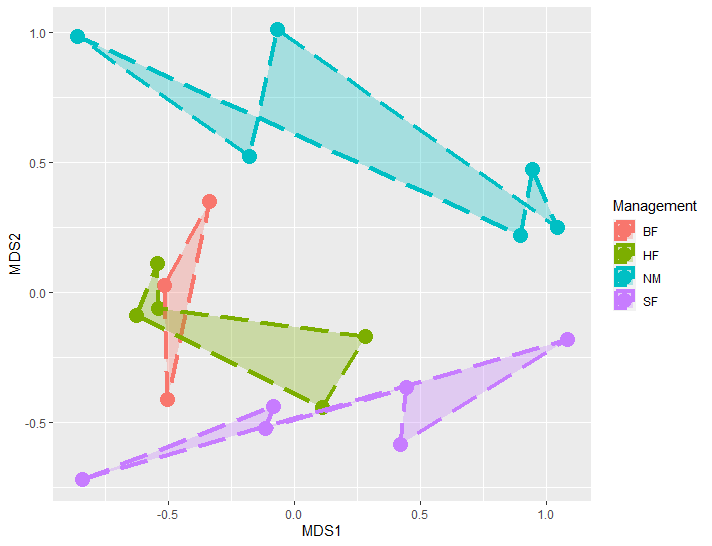
07
由于geom_polygon()会按照数据中出现的顺序连接观测值,因此如果我们按照df自身顺序来绘制多边形,多边形会非常奇怪,没法代表不同分组。因此,我们需要预先处理df的顺序,按合理的顺序连接观测值。
df <- df[order(df$Management), ]#先按分组排序 df$Order <- c(2, 1, 3, 1, 2, 3, 4, 5, 3, 5, 1, 6, 2, 4, 1, 2, 6, 3, 5, 4)#添加一列Order,给每个分组内观测点的手动排序 df <- df[order(df$Management, df$Order), ]#按分组和Order排序 p <- ggplot(df, aes(MDS1, MDS2))+ geom_point(aes(color = Management), size = 5)+ geom_polygon(aes(x = MDS1, y = MDS2, fill = Management, group = Management, color = Management), alpha = 0.3, linetype = "longdash", linewidth = 1.5)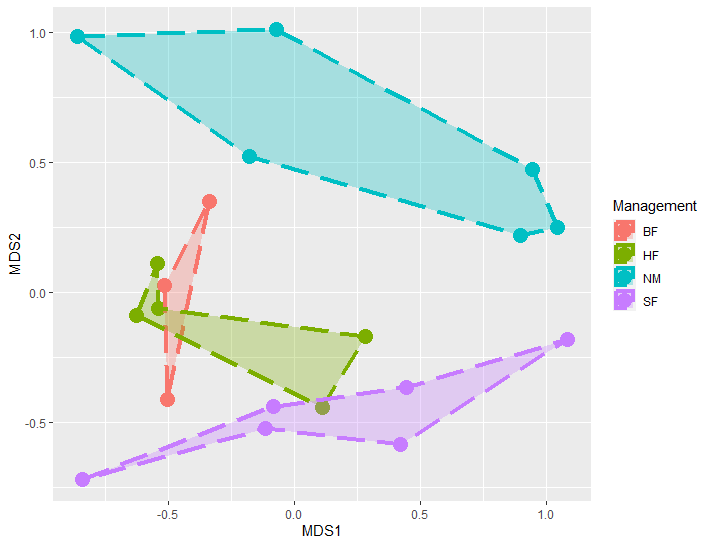
08
分别进行Anosim分析(Analysis of similarities)和PERMANOVA(即adonis)检验分析。
讯享网#设置随机种子 set.seed(123) #基于bray-curtis距离进行PERMANOVA分析 adonis <- adonis2(dune ~ Management, data = dune.env, permutations = 999, method = "bray") #基于bray-curtis距离进行anosim分析 anosim = anosim(dune, dune.env$Management, permutations = 999, distance = "bray")
09
美化图片,并用AI微调。
# 应力值stress,Adonis R2与显著性,Anosim R与显著性 stress_text <- paste("Stress =", round(stress, 4)) adonis_text <- paste(paste("Adonis =", round(adonis$R2, 2)), "")[1] anosim_text <- paste(paste("Anosim =", round(anosim$statistic, 2)), "") p <- ggplot(df, aes(MDS1, MDS2))+ geom_point(aes(color = Management), size = 5)+ geom_polygon(aes(x = MDS1, y = MDS2, fill = Management, group = Management, color = Management), alpha = 0.3, linetype = "longdash", linewidth = 1.5)+ theme(plot.margin = unit(rep(1, 4), 'lines'), panel.border = element_rect(fill = NA, color = "black", size = 0.5, linetype = "solid"), panel.grid = element_blank(), panel.background = element_rect(fill = 'white'))+ guides(color = "none", fill = "none")+ ggtitle(paste(paste(stress_text, adonis_text), anosim_text))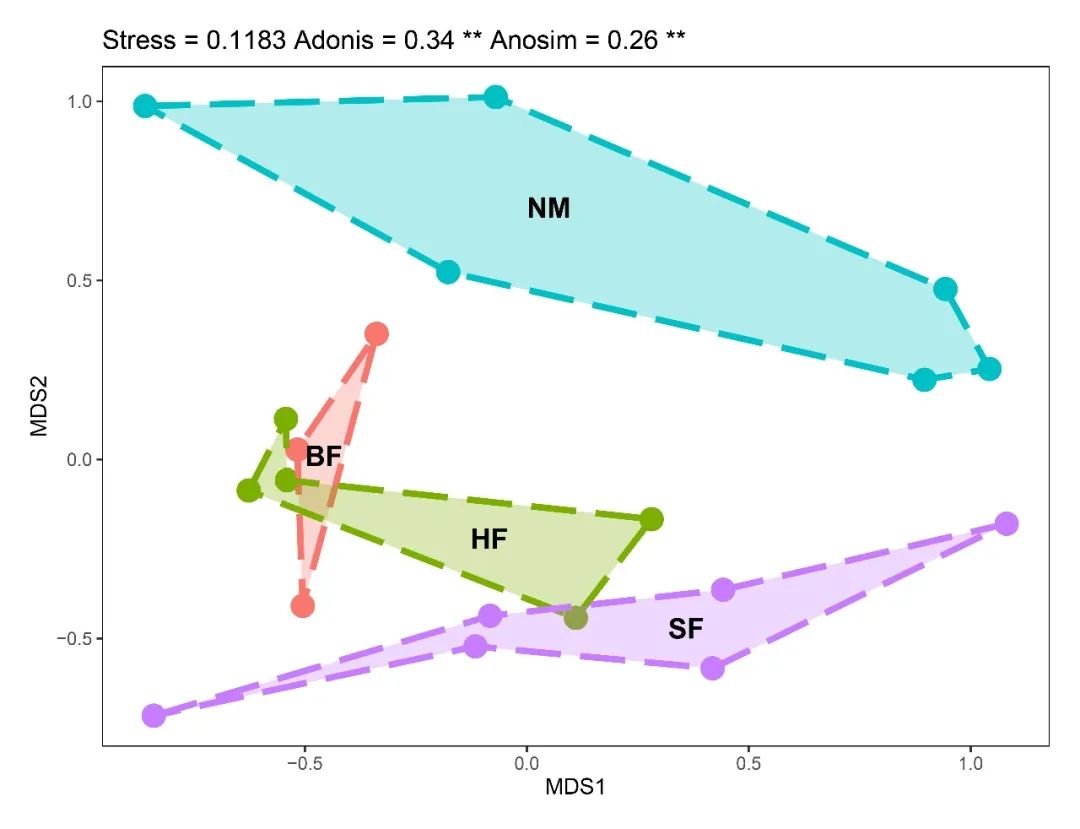
完整代码
讯享网if (!require("vegan")) install.packages('vegan') if (!require("ggplot2")) install.packages('ggplot2') # 加载包 library(vegan) library(ggplot2) # 载入dune数据集 data(dune) #载入dune包含分组信息等的元数据(即metadata),分组信息为Management列 data(dune.env) #计算bray_curtis距离 distance <- vegdist(dune, method = 'bray') #NMDS排序分析,k = 2预设两个排序轴 nmds <- metaMDS(distance, k = 2) #> Run 0 stress 0. #> Run 1 stress 0. #> ... Procrustes: rmse 1.e-05 max resid 4.e-05 #> ... Similar to previous best #> Run 2 stress 0. #> ... Procrustes: rmse 3.e-06 max resid 1.009651e-05 #> ... Similar to previous best #> Run 3 stress 0. #> Run 4 stress 0. #> ... Procrustes: rmse 0.000 max resid 0.000 #> ... Similar to previous best #> Run 5 stress 0. #> Run 6 stress 0. #> Run 7 stress 0. #> ... Procrustes: rmse 1.e-05 max resid 4.e-05 #> ... Similar to previous best #> Run 8 stress 0. #> Run 9 stress 0. #> ... Procrustes: rmse 1.e-05 max resid 5.e-05 #> ... Similar to previous best #> Run 10 stress 0. #> ... Procrustes: rmse 6.e-06 max resid 1.e-05 #> ... Similar to previous best #> Run 11 stress 0. #> ... Procrustes: rmse 5.e-05 max resid 0.000 #> ... Similar to previous best #> Run 12 stress 0. #> ... New best solution #> ... Procrustes: rmse 5.074111e-06 max resid 1.e-05 #> ... Similar to previous best #> Run 13 stress 0. #> ... Procrustes: rmse 3.e-05 max resid 9.85043e-05 #> ... Similar to previous best #> Run 14 stress 0. #> Run 15 stress 0. #> Run 16 stress 0. #> Run 17 stress 0. #> ... Procrustes: rmse 2.e-05 max resid 7.079487e-05 #> ... Similar to previous best #> Run 18 stress 0. #> ... New best solution #> ... Procrustes: rmse 0.0 max resid 0.0 #> Run 19 stress 0. #> ... New best solution #> ... Procrustes: rmse 3.78469e-06 max resid 9.e-06 #> ... Similar to previous best #> Run 20 stress 0. #> * Best solution repeated 1 times #查看结果 #summary(nmds) #获得应力值(stress) stress <- nmds$stress #将绘图数据转化为数据框 df <- as.data.frame(nmds$points) #与分组数据合并 df <- cbind(df, dune.env) df <- df[order(df$Management), ]#先按分组排序 df$Order <- c(2, 1, 3, 1, 2, 3, 4, 5, 3, 5, 1, 6, 2, 4, 1, 2, 6, 3, 5, 4)#添加一列Order,给每个分组内观测点的手动排序 df <- df[order(df$Management, df$Order), ]#按分组和Order排序 #设置随机种子 set.seed(123) #基于bray-curtis距离进行PERMANOVA分析 adonis <- adonis2(dune ~ Management, data = dune.env, permutations = 999, method = "bray") #基于bray-curtis距离进行anosim分析 anosim = anosim(dune, dune.env$Management, permutations = 999, distance = "bray") # 应力值stress,Adonis R2与显著性,Anosim R与显著性 stress_text <- paste("Stress =", round(stress, 4)) adonis_text <- paste(paste("Adonis =", round(adonis$R2, 2)), "")[1] anosim_text <- paste(paste("Anosim =", round(anosim$statistic, 2)), "") p <- ggplot(df, aes(MDS1, MDS2))+ geom_point(aes(color = Management), size = 5)+ geom_polygon(aes(x = MDS1, y = MDS2, fill = Management, group = Management, color = Management), alpha = 0.3, linetype = "longdash", linewidth = 1.5)+ theme(plot.margin = unit(rep(1, 4), 'lines'), panel.border = element_rect(fill = NA, color = "black", size = 0.5, linetype = "solid"), panel.grid = element_blank(), panel.background = element_rect(fill = 'white'))+ guides(color = "none", fill = "none")+ ggtitle(paste(paste(stress_text, adonis_text), anosim_text)) ggsave("Figure6C.pdf", p, height = 5.69, width = 7.42)
猜你喜欢
iMeta简介 高引文章 高颜值绘图imageGP 网络分析iNAP
iMeta网页工具 代谢组MetOrigin 美吉云乳酸化预测DeepKla
iMeta综述 肠菌菌群 植物菌群 口腔菌群 蛋白质结构预测
10000+:菌群分析 宝宝与猫狗 梅毒狂想曲 提DNA发Nature
系列教程:微生物组入门 Biostar 微生物组 宏基因组
专业技能:学术图表 高分文章 生信宝典 不可或缺的人
一文读懂:宏基因组 寄生虫益处 进化树 必备技能:提问 搜索 Endnote
扩增子分析:图表解读 分析流程 统计绘图
16S功能预测 PICRUSt FAPROTAX Bugbase Tax4Fun
生物科普: 肠道细菌 人体上的生命 生命大跃进 细胞暗战 人体奥秘
写在后面
为鼓励读者交流快速解决科研困难,我们建立了“宏基因组”讨论群,己有国内外6000+ 科研人员加入。请添加主编微信meta-genomics带你入群,务必备注“姓名-单位-研究方向-职称/年级”。高级职称请注明身份,另有海内外微生物PI群供大佬合作交流。技术问题寻求帮助,首先阅读《如何优雅的提问》学习解决问题思路,仍未解决群内讨论,问题不私聊,帮助同行。
点击阅读原文,跳转最新文章目录阅读


版权声明:本文内容由互联网用户自发贡献,该文观点仅代表作者本人。本站仅提供信息存储空间服务,不拥有所有权,不承担相关法律责任。如发现本站有涉嫌侵权/违法违规的内容,请联系我们,一经查实,本站将立刻删除。
如需转载请保留出处:https://51itzy.com/kjqy/28197.html