一.找目的基因的CDS序列保守区域
通过Nucleotide数据库查询目的基因
进入NCBI的Nucleotide数据库:
https://www.ncbi.nlm.nih.gov/nucleotide/
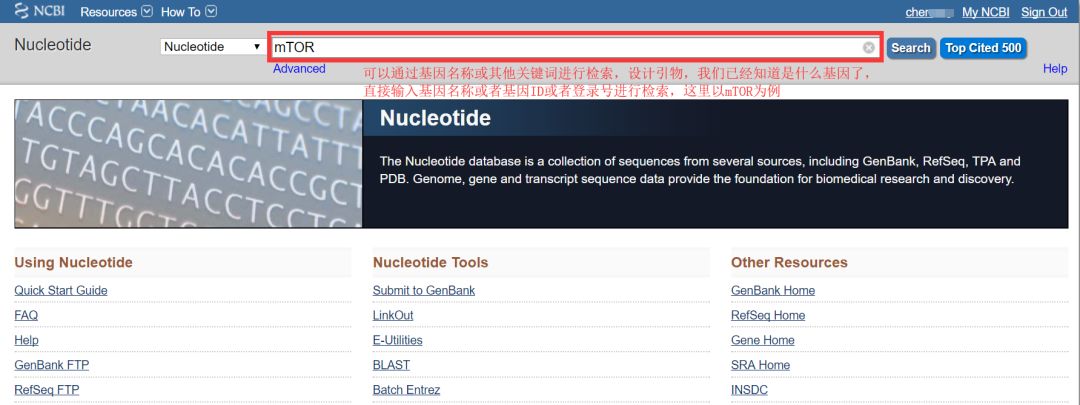
在搜索框直接键入要搜索的基因,这里以人的mTOR基因为例,检索结果如下:
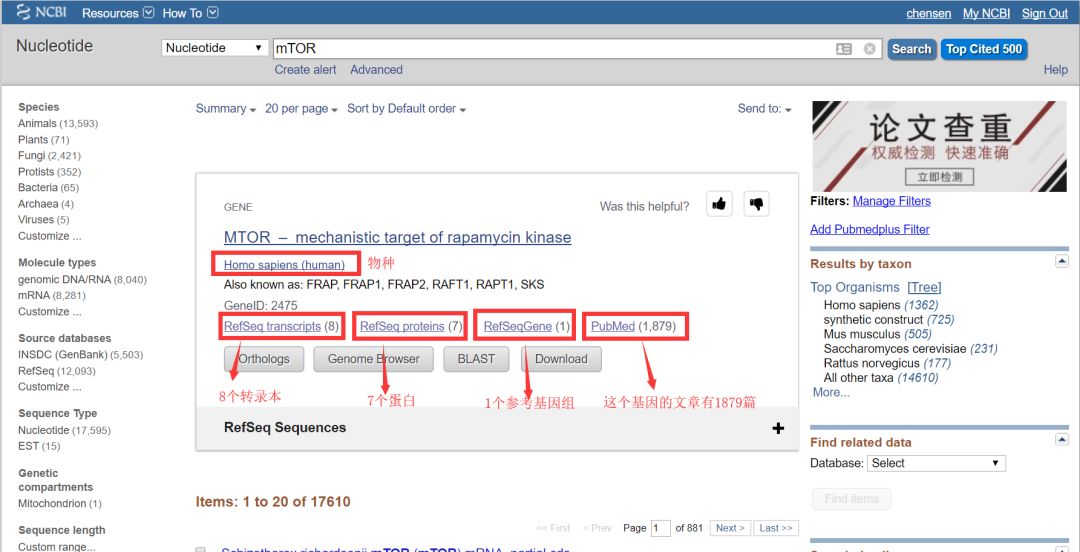
Nucleotide数据库的搜索结果还是比较智能的,搜索结果前面推荐的就是我们的要的,我们做基因检测,做的PCR是定量PCR,设计引物是以基因的转录本为模板设计的,所以我们点击RefSeq transcripts。
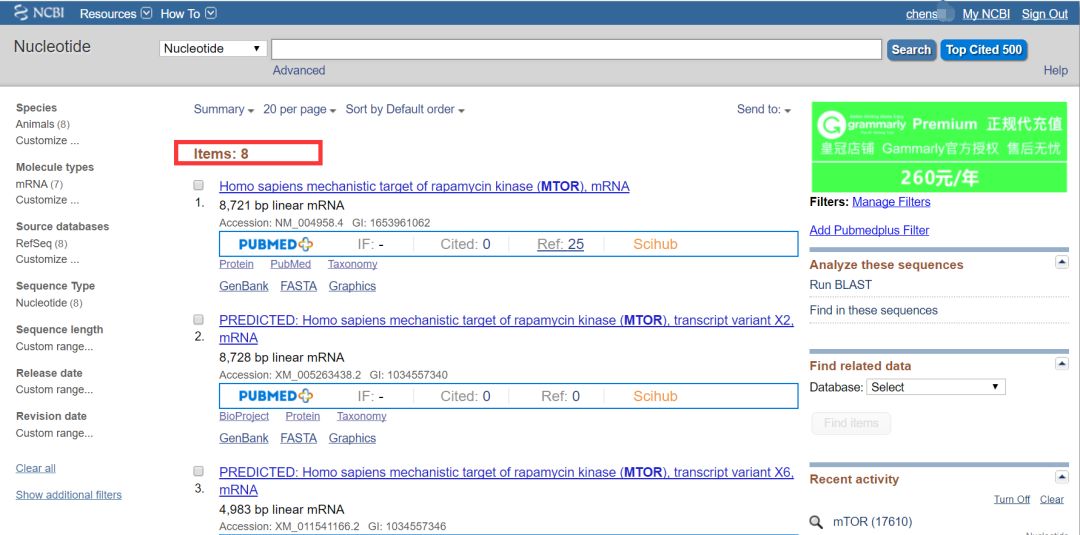
随便点击其中一个
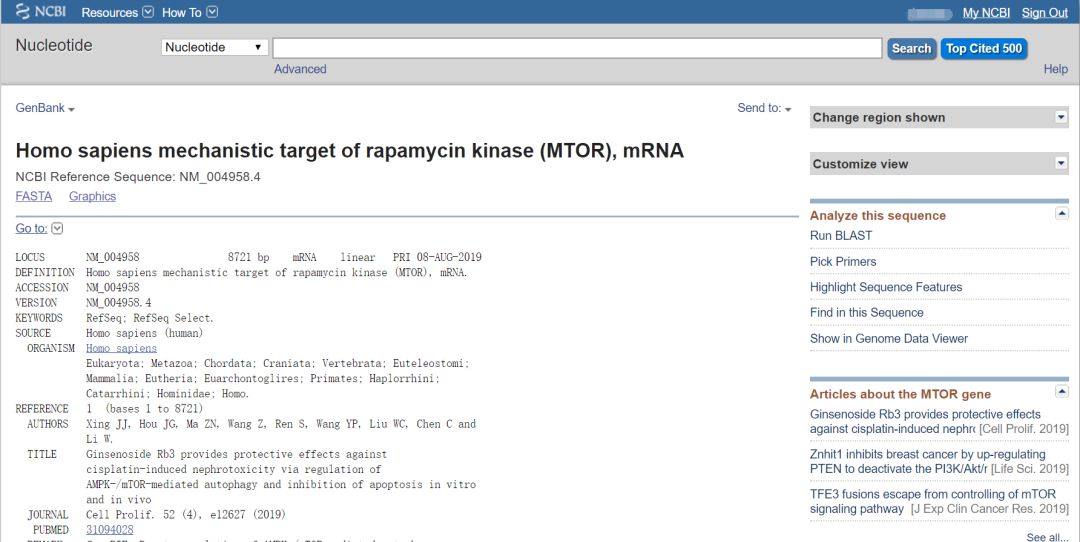
该页面的信息想了解的可参考文章:生物信息学初识篇——第一章:生物数据库,这里我们只需要往下拉,找到CDS。
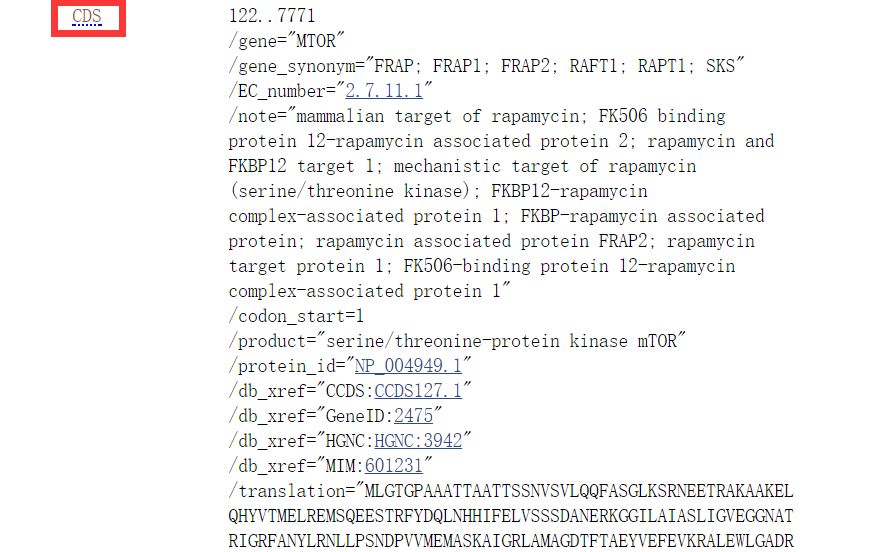
点击CDS,页面会跳到序列位置,并将CDS区背景高亮。

将序列以fasta格式粘贴到一个Word文档中

同样的方法将其他转录本的CDS序列也复制粘贴进来。这里需要注意的是,8个转录本中,有7个是软件预测的,只有第一个是被验证过的。设计引物时可以只用已经验证的这一个,如果其他基因,有多个已经验证转录本,就抛弃那些软件预测的序列,只要验证的序列。这里的mTOR只有一个被验证,理论上可以只有这一条序列设计引物,这里为了演示,我也下载了其他预测的序列进行了序列比对。
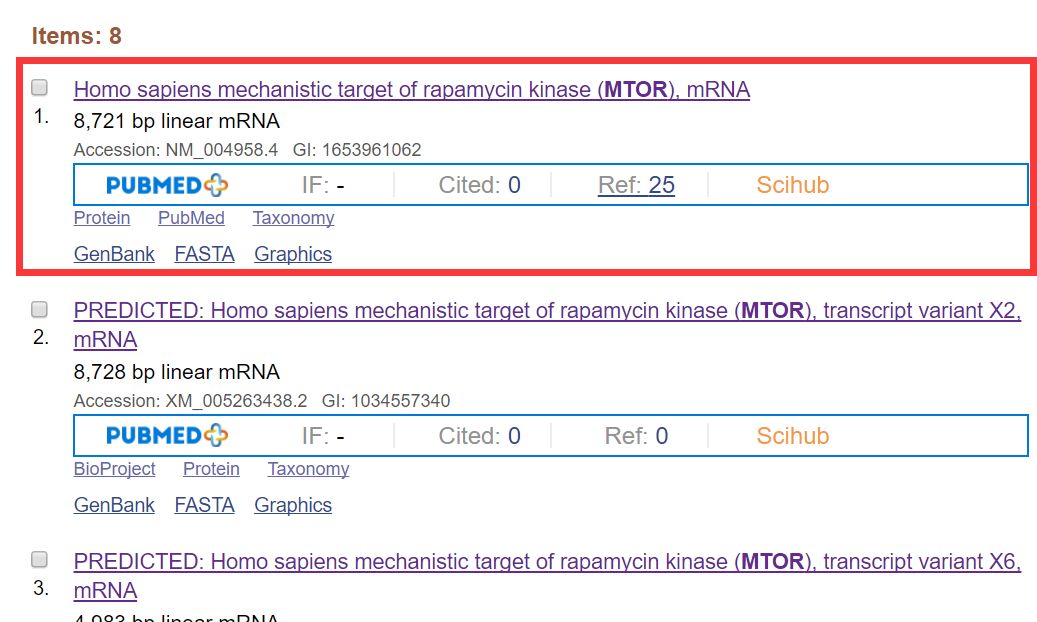
将所有序列都以fasta格式粘贴到同一个word文档中后,利用Word的查找替换功能将所有数字删掉。
然后利用MEGA 7 软件进行序列比对,找到7个转录本的保守区域,关于MEGA 7 软件使用教程和序列比对,可参考:生物信息学初识篇——第三章:分子进化与系统发生,该文章介绍了MEGA 7建系统发育树的过程,案例是做蛋白,只需要将蛋白序列换成DNA序列即可。序列比对部分是一样的,只是不需要用来建系统发育树,或者自己百度一下MEGA 7做序列比对,教程应该很多,其实做序列比对的工具很多,之前的文章也有介绍,可以参考本公众号的文章:生物信息学初识篇——第二章的内容。
我们通过比对后获得mTOR的保守区域序列:
ATGAACCATGTCCTAAGCTGTGTCAAGAAGGAGAAGGAACGTACAGCGGCCTTCCAAGCCCTGGGGCTACTTTCTGTGGCTGTGAGGTCTGAGTTTAAGGTCTATTTGCCTCGCGTGCTGGACATCATCCGAGCGGCCCTGCCCCCAAAGGACTTCGCCCATAAGAGGCAGAAGGCAATGCAGGTGGATGCCACAGTCTTCACTTGCATCAGCATGCTGGCTCGAGCAATGGGGCCAGGCATCCAGCAGGATATCAAGGAGCTGCTGGAGCCCATGCTGGCAGTGGGACTAAGCCCTGCCCTCACTGCAGTGCTCTACGACCTGAGCCGTCAGATTCCACAGCTAAAGAAGGACATTCAAGATGGGCTACTGAAAATGCTGTCCCTGGTCCTTATGCACAAACCCCTTCGCCACCCAGGCATGCCCAAGGGCCTGGCCCATCAGCTGGCCTCTCCTGGCCTCACGACCCTCCCTGAGGCCAGCGATGTGGGCAGCATCACTCTTGCCCTCCGAACGCTTGGCAGCTTTGAATTTGAAGGCCACTCTCTGACCCAATTTGTTCGCCACTGTGCGGATCATTTCCTGAACAGTGAGCACAAGGAGATCCGCATGGAGGCTGCCCGCACCTGCTCCCGCCTGCTCACACCCTCCATCCACCTCATCAGTGGCCATGCTCATGTGGTTAGCCAGACCGCAGTGCAAGTGGTGGCAGATGTGCTTAGCAAACTGCTCGTAGTTGGGATAACAGATCCTGACCCTGACATTCGCTACTGTGTCTTGGCGTCCCTGGACGAGCGCTTTGATGCACACCTGGCCCAGGCGGAGAACTTGCAGGCCTTGTTTGTGGCTCTGAATGACCAGGTGTTTGAGATCCGGGAGCTGGCCATCTGCACTGTGGGCCGACTCAGTAGCATGAACCCTGCCTTTGTCATGCCTTTCCTGCGCAAGATGCTCATCCAGATTTTGACAGAGTTGGAGCACAGTGGGATTGGAAGAATCAAAGAGCAGAGTGCCCGCATGCTGGGGCACCTGGTCTCCAATGCCCCCCGACTCATCCGCCCCTACATGGAGCCTATTCTGAAGGCATTAATTTTGAAACTGAAAGATCCAGACCCTGATCCAAACCCAGGTGTGATCAATAATGTCCTGGCAACAATAGGAGAATTGGCACAGGTTAGTGGCCTGGAAATGAGGAAATGGGTTGATGAACTTTTTATTATCATCATGGACATGCTCCAGGATTCCTCTTTGTTGGCCAAAAGGCAGGTGGCTCTGTGGACCCTGGGACAGTTGGTGGCCAGCACTGGCTATGTAGTAGAGCCCTACAGGAAGTACCCTACTTTGCTTGAGGTGCTACTGAATTTTCTGAAGACTGAGCAGAACCAGGGTACACGCAGAGAGGCCATCCGTGTGTTAGGGCTTTTAGGGGCTTTGGATCCTTACAAGCACAAAGTGAACATTGGCATGATAGACCAGTCCCGGGATGCCTCTGCTGTCAGCCTGTCAGAATCCAAGTCAAGTCAGGATTCCTCTGACTATAGCACTAGTGAAATGCTGGTCAACATGGGAAACTTGCCTCTGGATGAGTTCTACCCAGCTGTGTCCATGGTGGCCCTGATGCGGATCTTCCGAGACCAGTCACTCTCTCATCATCACACCATGGTTGTCCAGGCCATCACCTTCATCTTCAAGTCCCTGGGACTCAAATGTGTGCAGTTCCTGCCCCAGGTCATGCCCACGTTCCTTAACGTCATTCGAGTCTGTGATGGGGCCATCCGGGAATTTTTGTTCCAGCAGCTGGGAATGTTGGTGTCCTTTGTGAAGAGCCACATCAGACCTTATATGGATGAAATAGTCACCCTCATGAGAGAATTCTGGGTCATGAACACCTCAATTCAGAGCACGATCATTCTTCTCATTGAGCAAATTGTGGTAGCTCTTGGGGGTGAATTTAAGCTCTACCTGCCCCAGCTGATCCCACACATGCTGCGTGTCTTCATGCATGACAACAGCCCAGGCCGCATTGTCTCTATCAAGTTACTGGCTGCAATCCAGCTGTTTGGCGCCAACCTGGATGACTACCTGCATTTACTGCTGCCTCCTATTGTTAAGTTGTTTGATGCCCCTGAAGCTCCACTGCCATCTCGAAAGGCAGCGCTAGAGACTGTGGACCGCCTGACGGAGTCCCTGGATTTCACTGACTATGCCTCCCGGATCATTCACCCTATTGTTCGAACACTGGACCAGAGCCCAGAACTGCGCTCCACAGCCATGGACACGCTGTCTTCACTTGTTTTTCAGCTGGGGAAGAAGTACCAAATTTTCATTCCAATGGTGAATAAAGTTCTGGTGCGACACCGAATCAATCATCAGCGCTATGATGTGCTCATCTGCAGAATTGTCAAGGGATACACACTTGCTGATGAAGAGGAGGATCCTTTGATTTACCAGCATCGGATGCTTAGGAGTGGCCAAGGGGATGCATTGGCTAGTGGACCAGTGGAAACAGGACCCATGAAGAAACTGCACGTCAGCACCATCAACCTCCAAAAGGCCTGGGGCGCTGCCAGGAGGGTCTCCAAAGATGACTGGCTGGAATGGCTGAGACGGCTGAGCCTGGAGCTGCTGAAGGACTCATCATCGCCCTCCCTGCGCTCCTGCTGGGCCCTGGCACAGGCCTACAACCCGATGGCCAGGGATCTCTTCAATGCTGCATTTGTGTCCTGCTGGTCTGAACTGAATGAAGATCAACAGGATGAGCTCATCAGAAGCATCGAGTTGGCCCTCACCTCACAAGACATCGCTGAAGTCACACAGACCCTCTTAAACTTGGCTGAATTCATGGAACACAGTGACAAGGGCCCCCTGCCACTGAGAGATGACAATGGCATTGTTCTGCTGGGTGAGAGAGCTGCCAAGTGCCGAGCATATGCCAAAGCACTACACTACAAAGAACTGGAGTTCCAGAAAGGCCCCACCCCTGCCATTCTAGAATCTCTCATCAG
二.设计引物
引物选择的一般原则:
避免多个重复碱基出现,避免4个或超过4个G出现在引物序列中;按照PCR引物要求设置参数,如产物长度,荧光定量PCR产物不应该超过400bp,最好80-150bp, 普通的PCR产物长度在300-400bp左右为宜;GC%为30%-70%,最好45%-55%;引物长度为18-25bp,最好22-24bp;Tm值在58-60℃,最好60℃;3’端至少4个碱基保守,最好为T, C, G;
荧光定量PCR的引物尽量选择G、C结尾的引物。
设计引物的工具很多,这里以Primer Premier 6.0为例。
Primer Premier 6.0安装包:
链接:https://pan.baidu.com/s/11vack5Z0x5nUeWQkYjWdNg 提取码:ac27
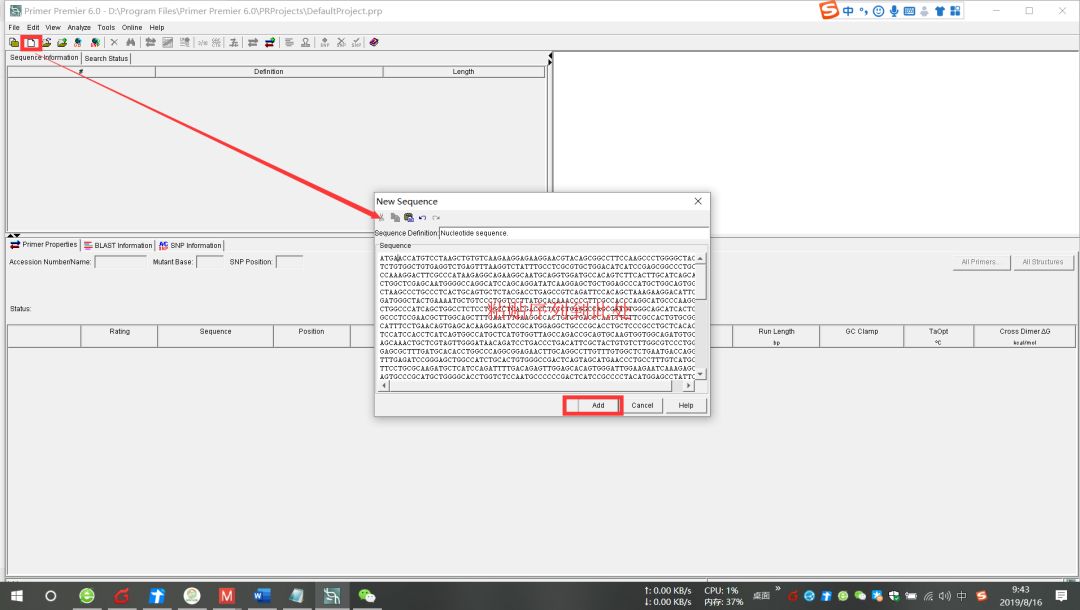
打开软件Primer Premier 6.0,点击左上角的New sequence,复制找到的保守区序列,到图中位置,点击Add。
点primer search,设置引物参数
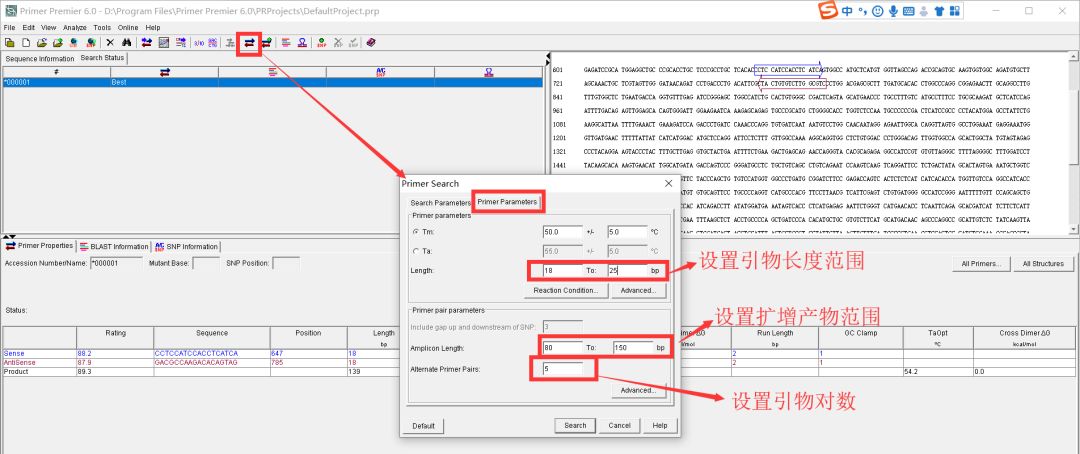
点击Search,下面显示所有结果中最优的一对引物,从各种参数来看,这对引物质量不错。

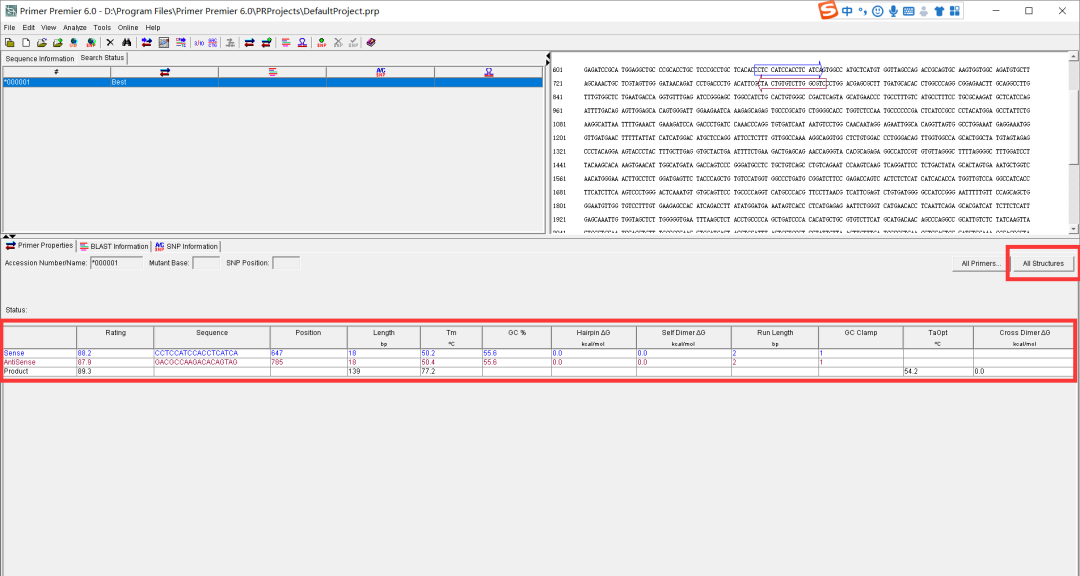
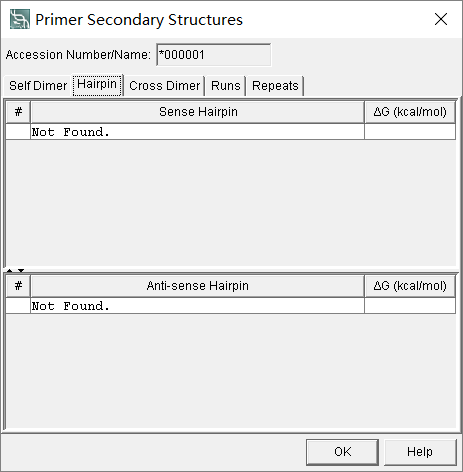
更多的引物参数还需要点击All Structures进行查看,比如是否形成发夹结构,可以看到这对引物不会形成发夹结构。
所以我们就获得了人mTOR基因的mRNA定量PCR的引物:
上游引物F:CCTCCATCCACCTCATCA
下游引物R:GACGCCAAGACACAGTAG
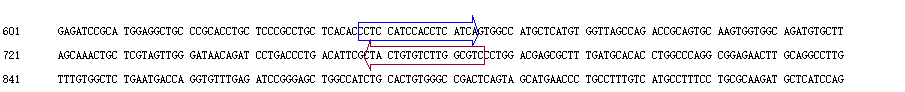
CTACTGTGTCTTGGCGTC
三.引物验证
NCBI BLAST网址http://blast.ncbi.nlm.nih.gov/Blast.cgi,点击Nucleotide
BLAST
在打开的界面中Enter Query Sequence下面框中,按照5’-3’顺序,分别输入上游引物和下游引物,上下游引物之间间隔20个以上n。
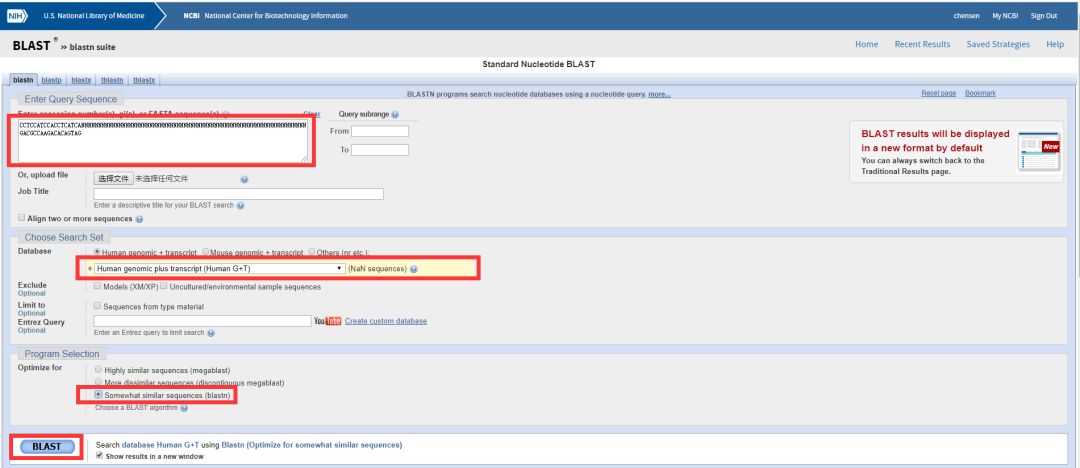
Program Selection选择Somewhat similar sequences(blastn) ,点击BLAST
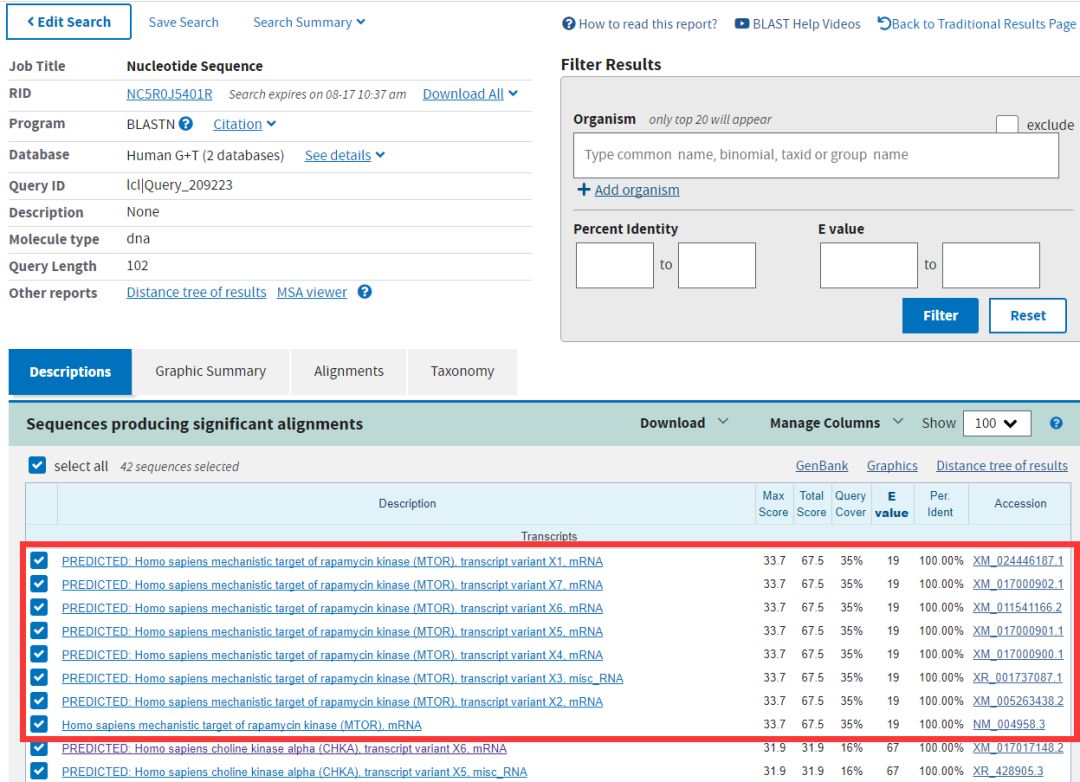
可以看到搜索结果排在前面的就是我们的mTOR,我们就可以找公司合成,然后进行实验,在实验中进一步验证,如果引物的扩增曲线具有单一的峰,说明引物设计没有问题。
四.其他引物设计工具
1. Thermo Fisher的Multiple Primer Analyzer
https://www.thermofisher.com/cn/zh/home/brands/thermo-scientific/molecular-biology/molecular-biology-learning-center/molecular-biology-resource-library/thermo-scientific-web-tools/multiple-primer-analyzer.html
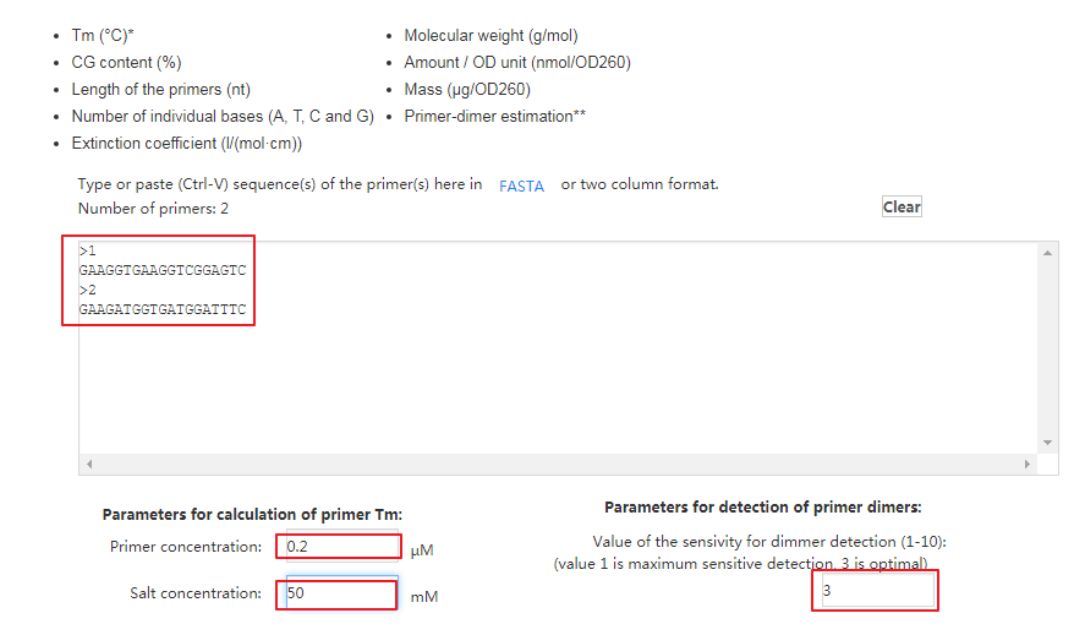
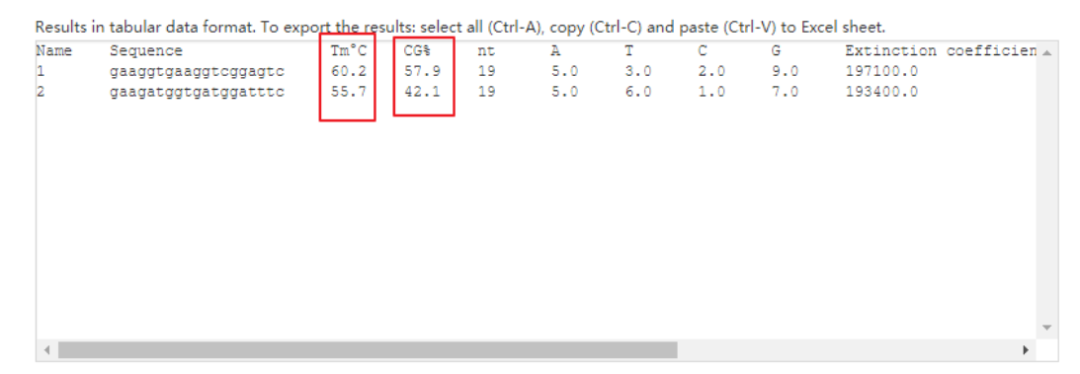
注意:如上图的格式输入上下游引物序列,还要确定引物的工作浓度、反应体系的盐浓度、评价的灵敏性。结果如下图。得到预查询引物的TM值, GC含量以及是否形成引物二聚体。
2.Primer3web在线引物设计
http://primer3.ut.ee/
3.Universal Probe Library在线引物设计
https://lifescience.roche.com/en_cn/brands/universal-probelibrary.html#assay-design-center
4.最后给两个小工具,评价单条引物
不过要注意这里给出的TM值为跑PCR 时推荐设定的退火温度。
http://www.ab126.com/Chemistry/3160.html
http://ggene.cn/html/protocol/bioinformatics/2015/0415/6838.html
相关文章
1.一文解决Western Blotting实验所有问题
2.TRlzol法提取组织或细胞RNA
3.反转录PCR
4.基因克隆有这篇文章就够了
5.基因过表达——融合基因过表达



版权声明:本文内容由互联网用户自发贡献,该文观点仅代表作者本人。本站仅提供信息存储空间服务,不拥有所有权,不承担相关法律责任。如发现本站有涉嫌侵权/违法违规的内容,请联系我们,一经查实,本站将立刻删除。
如需转载请保留出处:https://51itzy.com/kjqy/11728.html